一般的富集气泡图最多可以展示四个维度的信息,如常见的同时展示pathway(y轴)、count(气泡大小)、p value(映射颜色)、gene ratio(x轴)。
桑基图常用于展示关联数据间的流动,在气泡图左侧通过添加桑基图,可将每个pathway中count对应的geneID直观展示,从而实现五个维度富集信息的展现,如下图两个文献中的案例:
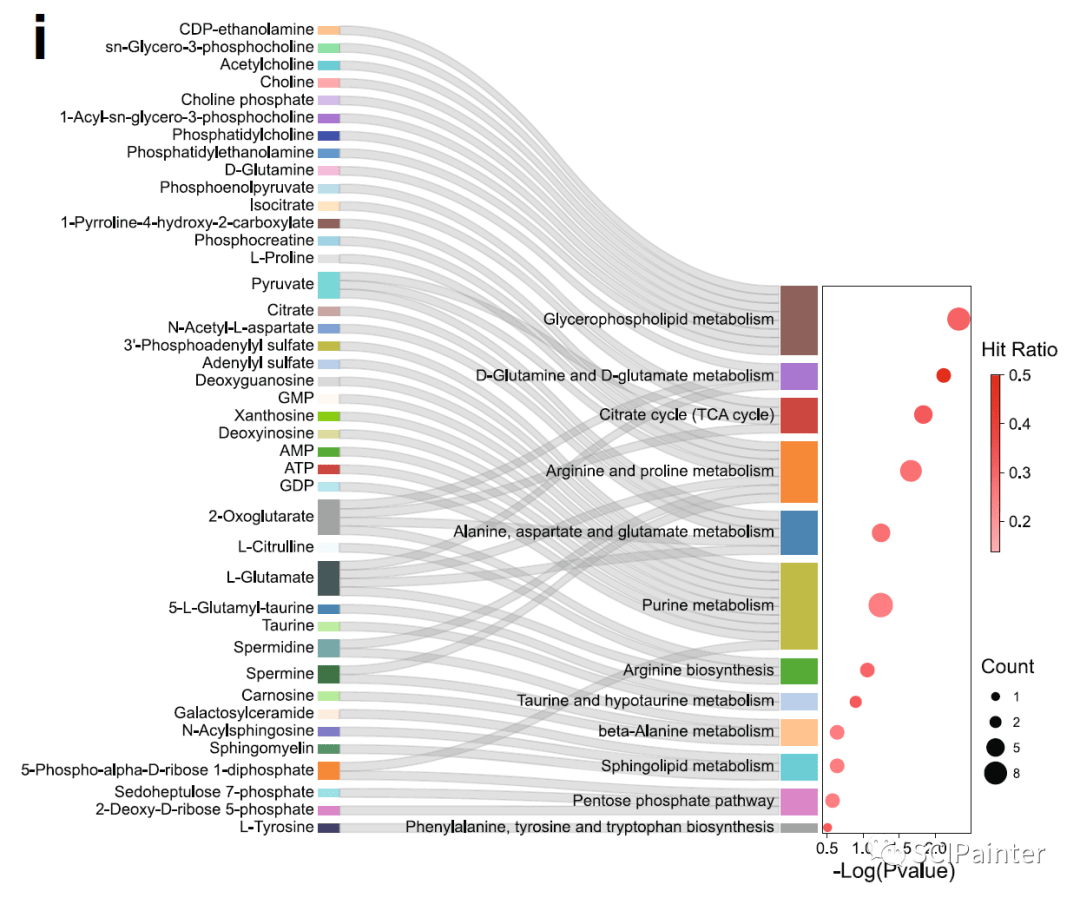
( Nat Commun ,2022)
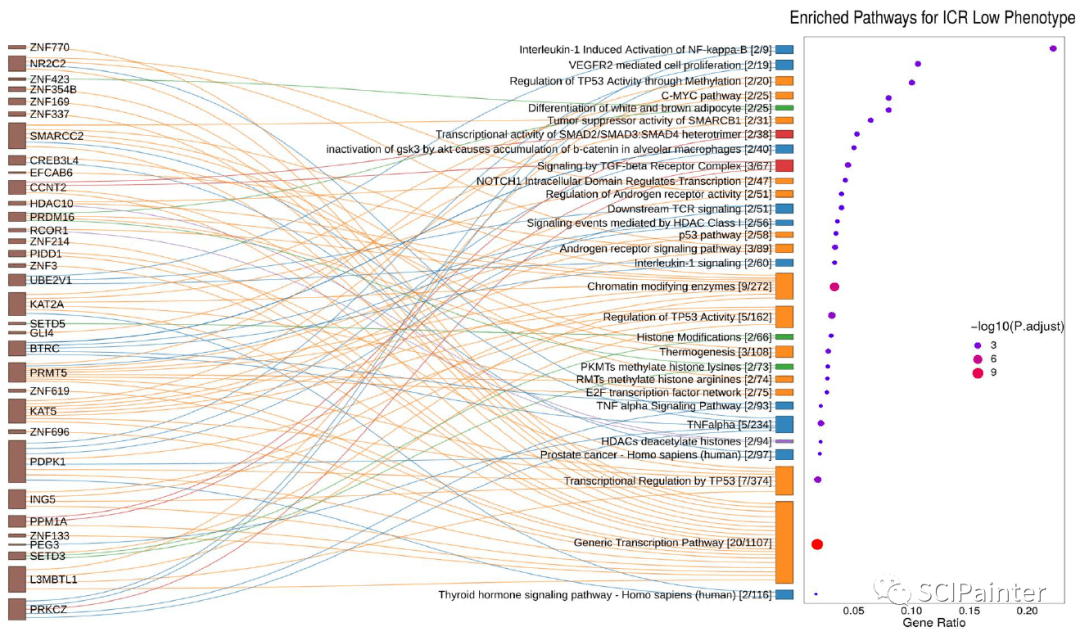
( Briefings in Bioinformatics ,2021)
基本绘图逻辑为先分别绘制富集气泡图和桑基图,然后进行拼接。话不多说,下面就来一起学习如何绘制吧!
#相关R包载入:
library(tidyverse)
library(ggsankey)
library(ggplot2)
library(cols4all)
library(cowplot)
#本地数据载入:
##富集气泡图数据:
kegg<- read.csv('enrichdt.csv',header = T)
head(kegg)
##桑基图数据:
sankey<- read.csv('sankeydt.csv',header = T)
head(sankey)
1. 富集气泡图绘制
#如果我们按照以往常规的绘制方法:
#指定绘图顺序(转换为因子):
kegg$pathNames <- factor(kegg$pathNames,levels = rev(kegg$pathNames))
#基础富集气泡图:
p1<- ggplot +
geom_point(data = kegg,
aes(x = -log10(Pvalue),
y= pathNames,
size= count,
color= Hit.Ratio)) + # 气泡大小及颜色设置
theme_bw+
labs(x = "-log10(Pvalue)",
y= "")
p1

这样绘制出来的图表y轴间距,也就是我们标签间的距离是相同的,但我们的组合图表中需要间距不等(根据count的比例来决定间距),这样才能和桑基图对应拼接。
这里可以参考的一种处理方式为:将原本的y轴标签(分类型变量)更改为数值型标签,从而实现不等距的效果。
#数据处理:
kegg2<- kegg[12:1,] #先调转数据框方向
kegg2<- kegg2 %>%
mutate(ymax = cumsum(count)) %>% #ymax为Width列的累加和
mutate(ymin = ymax -count) %>%
mutate(label = (ymin + ymax)/2) #取xmin和xmax的中心位置作为标签位置
head(kegg2)
p2<- ggplot +
geom_point(data = kegg2,
aes(x = -log10(Pvalue),
y= label, #替换y轴列为数值
size= count,
color= Hit.Ratio)) +
theme_bw+
labs(x = "-log10(Pvalue)",
y= "")
p2

#自定义主题与配色修改:
mytheme<- theme(axis.title = element_text(size = 13),
axis.text = element_text(size = 11),
axis.text.y = element_blank,
axis.ticks.y = element_blank,
legend.title = element_text(size = 13),
legend.text = element_text(size = 11))
p3<- ggplot +
geom_point(data = kegg2,
aes(x = -log10(Pvalue),
y= label,
size= count,
color= Hit.Ratio)) +
scale_size_continuous(range=c(2,8)) + #调整气泡大小范围(默认尺寸部分过小)
scale_y_continuous(expand = c(0,0.1),limits = c(0,52)) +
scale_x_continuous(limits = c(0.57,2.5)) +
scale_colour_distiller(palette = "Reds", direction = 1) + #更改配色
labs(x = "-log10(Pvalue)",
y= "") +
theme_bw+
mytheme
p3

现在用于拼接的富集气泡图就画好啦!下面我们使用ggsankey包绘制桑基图,该R包的详细使用方法可戳往期:《如何绘制高分文献的同款桑基图?炫酷+轻松=完胜!》
2. 桑基图绘制
#将数据转换为绘图所需格式:
df<- sankey %>%
make_long(metamolites, pathNames)
head(df)
#指定绘图顺序(转换为因子):
df$node <- factor(df$node,levels = c(sankey$pathNames %>% unique%>% rev,
sankey$metamolites %>% unique %>% rev))
#自定义配色:
c4a_gui
mycol<- c4a('rainbow_wh_rd',53)
#绘图:
p4<- ggplot(df, aes(x = x,
next_x= next_x,
node= node,
next_node= next_node,
fill= node,
label= node)) +
geom_sankey(flow.alpha = 0.5,
flow.fill = 'grey',
flow.color = 'grey80', #条带描边色
node.fill = mycol, #节点填充色
smooth= 8,
width= 0.08) +
geom_sankey_text(size = 3.2,
color= "black")+
theme_void+
theme(legend.position = 'none')
p4

3. 拼图
#通过调整页边距在桑基图右侧先留出空白:
p5<- p4 + theme(plot.margin = unit(c(0,5,0,0),units= "cm"))
p5

#拼图(在p5的空白位置中插入p3):
ggdraw+ draw_plot(p5) + draw_plot(p3, scale = 0.5, x = 0.62, y=-0.21, width=0.48, height=1.37)

具体的位置需要根据实际反复调整,直至对齐。到这里我们基本完成了桑基气泡组合图的绘制,如果想要精益求精,可以最后在AI中调整一下标签的位置。
4. AI调整
在AI中打开我们上一步保存的图表(矢量格式),使用直接选择工具鼠标长按框选住桑基图右侧列的全部标签;

在右侧属性控制栏中选择对齐—水平右对齐,如果在右侧找不到,可以通过菜单栏窗口—对齐(shift+F7)调出该属性面板。

切换到选择工具,水平拖动标签到节点的左方即可。

桑基图左侧列标签的调整方法相同,我们就不再赘述啦。最后使用直接选择工具选中右侧的气泡图全部图形元素(包括文字和图例),再切换回选择工具,向左水平移动填满空白部分。

最终效果如下:

好啦,今天的分享就到这里!
【参考资料】
https://github.com/davidsjoberg/ggsankey
参考文献
Yu, W., Wang, Z., Yu, X. et al. Kir2.1-mediated membrane potential promotes nutrient acquisition and inflammation through regulation of nutrient transporters. Nat Commun 13, 3544 (2022).
Mall R , Saad M , Roelands J , et al. Network-based identification of key master regulators associated with an immune-silent cancer phenotype[J]. Briefings in bioinformatics:bbab168.
*未经许可,不得以任何方式复制或抄袭本篇文章之部分或全部内容。版权所有,侵权必究。
基迪奥生物|专业定制测序服务
联系方式:020-39341079;service@genedenovo.com返回搜狐,查看更多

